新网站 蜘蛛/找客户的十大方法
logistic回归又称logistic回归分析,是一种广义的线性回归分析模型,常用于数据挖掘,疾病自动诊断,经济预测等领域。例如,探讨引发疾病的危险因素,并根据危险因素预测疾病发生的概率等。以胃癌病情分析为例,选择两组人群,一组是胃癌组,一组是非胃癌组,两组人群必定具有不同的体征与生活方式等。因此因变量就为是否胃癌,值为“是”或“否”,自变量就可以包括很多了,如年龄、性别、饮食习惯、幽门螺杆菌感染等。自变量既可以是连续的,也可以是分类的。然后通过logistic回归分析,可以得到自变量的权重,从而可以大致了解到底哪些因素是胃癌的危险因素。同时根据该权值可以根据危险因素预测一个人患癌症的可能性。
——百度百科
目录
- 算法描述
- 算法简述
- Sigmoid函数
- 梯度
- 最优化方法确定最佳回归系数(梯度上升法)
- 优缺点
- 一般过程
- 一个栗子
- 从疝气病症预测病马的死亡率
算法描述
算法简述
Sigmoid函数
我们所需要的函数应该是,能接受所有的输入然后预测出类别,例如二分类,我们可以使用像Sigmoid这样的函数,它的图像如下,公式在图片右上角:

Sigmoid函数的特点是,两条渐近线y=1和y=0;当x大于0值越大,y值趋近于1;当x小于0值越小,y值趋近于0。这样的函数更方便于我们作分类问题,比如,我们设置预测结果y值大于0.5,就可以认为预测结果是类别1,y值小于0.5,就可以认为预测结果是类别0,当然,这些都可以自己去定义。除了Sigmoid,还可以使用Tanh,ReLU函数等,单击链接,本文使用的是Sigmoid函数。
梯度
定义Sigmoid的输入为z:

为了使得分类器尽可能精确,我们所需要的就是求出这些w值的最优结果,这里我们可以使用梯度上升法。
要学习梯度上升法至少要了解的先验知识:
- 偏导数
- 梯度
中学知识我们知道,导数是对一个函数进行求导计算,如果变量多于一个,那么对某一个变量进行求导计算,就称求它的偏导数,而梯度,就是将这些偏导数保存在一起,转为向量,所以梯度是一个向量值,向量中的每一个值分别是一个导数。这里是相关链接
梯度的值表示某一函数在该点处的方向导数沿着该方向取得最大值,想象一下,我们要求的w值是最优的,那么在空间中的函数图像,我们要求到的就是这个函数的最值。即下图中的红色圈出来的地方。

最优化方法确定最佳回归系数(梯度上升法)
使用梯度上升算法的迭代公式如下:
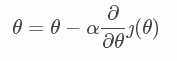
算法如下:
每个回归系数初始化为1
重复R次:计算整个数据集的梯度使用alpha × gradient更新回归系数的向量
返回回归系数
alpha是学习步长,也就是每次移动的大小,alpha过大可能会导致找不到最优解,过小可能会导致局部最优,所以找到合适的alpha也是受影响的。R是迭代次数,这里是算法是批量梯度算法,还有随机梯度和小批量梯度算法,分享个一个很棒的资料。
优缺点
- 优点:计算代价不高,易于理解
- 缺点:容易欠拟合,分类精度可能不高
- 适用数据类型:数值型、标称型
一般过程
- 收集数据:任意方法
- 准备数据:由于需要进行距离计算,因此要求数据类型为数值型。另外,结构化数据格式则最佳
- 分析数据:任意方法
- 训练算法:大部分时间将用于训练算法,目的是为了找到最佳的分类回归系数
- 测试算法:一旦训练步骤完成,分类将会很快
- 使用算法:首先,需要输入数据,将数据转换成对应的结构化数值;然后,基于训练好的回归系数就可以对这些数值进行简单的回归计算,判定是哪个类别;最后,我们可以在输出的类别上进行一些其他分析工作
一个栗子
from numpy import *def loadDataSet():dataMat = []; labelMat = []fr = open('testSet.txt')for line in fr.readlines():lineArr = line.strip().split()dataMat.append([1.0, float(lineArr[0]), float(lineArr[1])])labelMat.append(int(lineArr[2]))return dataMat,labelMatdef sigmoid(inX):return 1.0/(1+exp(-inX))def gradAscent(dataMatIn, classLabels):dataMatrix = mat(dataMatIn) #convert to NumPy matrixlabelMat = mat(classLabels).transpose() #convert to NumPy matrixm,n = shape(dataMatrix)alpha = 0.001maxCycles = 500weights = ones((n,1))for k in range(maxCycles): #heavy on matrix operationsh = sigmoid(dataMatrix*weights) #matrix multerror = (labelMat - h) #vector subtractionweights = weights + alpha * dataMatrix.transpose()* error #matrix multreturn weightsdef plotBestFit(weights):import matplotlib.pyplot as pltdataMat,labelMat=loadDataSet()dataArr = array(dataMat)n = shape(dataArr)[0] xcord1 = []; ycord1 = []xcord2 = []; ycord2 = []for i in range(n):if int(labelMat[i])== 1:xcord1.append(dataArr[i,1]); ycord1.append(dataArr[i,2])else:xcord2.append(dataArr[i,1]); ycord2.append(dataArr[i,2])fig = plt.figure()ax = fig.add_subplot(111)ax.scatter(xcord1, ycord1, s=30, c='red', marker='s')ax.scatter(xcord2, ycord2, s=30, c='green')x = arange(-3.0, 3.0, 0.1)y = (-weights[0]-weights[1]*x)/weights[2]ax.plot(x, y)plt.xlabel('X1'); plt.ylabel('X2');plt.show()def stocGradAscent0(dataMatrix, classLabels):m,n = shape(dataMatrix)alpha = 0.01weights = ones(n) #initialize to all onesfor i in range(m):h = sigmoid(sum(dataMatrix[i]*weights))error = classLabels[i] - hweights = weights + alpha * error * dataMatrix[i]return weightsdef stocGradAscent1(dataMatrix, classLabels, numIter=150):m,n = shape(dataMatrix)weights = ones(n) #initialize to all onesfor j in range(numIter):dataIndex = range(m)for i in range(m):alpha = 4/(1.0+j+i)+0.0001 #apha decreases with iteration, does not randIndex = int(random.uniform(0,len(dataIndex)))#go to 0 because of the constanth = sigmoid(sum(dataMatrix[randIndex]*weights))error = classLabels[randIndex] - hweights = weights + alpha * error * dataMatrix[randIndex]del(dataIndex[randIndex])return weightsdataArr,labelMat=loadDataSet()
weights=stocGradAscent1(array(dataArr),labelMat,500)
plotBestFit(weights)
结果:

从疝气病症预测病马的死亡率
def sigmoid(inX):return 1.0/(1+exp(-inX))def stocGradAscent1(dataMatrix, classLabels, numIter=150):m,n = shape(dataMatrix)weights = ones(n) #initialize to all onesfor j in range(numIter):dataIndex = range(m)for i in range(m):alpha = 4/(1.0+j+i)+0.0001 #apha decreases with iteration, does not randIndex = int(random.uniform(0,len(dataIndex)))#go to 0 because of the constanth = sigmoid(sum(dataMatrix[randIndex]*weights))error = classLabels[randIndex] - hweights = weights + alpha * error * dataMatrix[randIndex]del(dataIndex[randIndex])return weightsdef classifyVector(inX, weights):prob = sigmoid(sum(inX*weights))if prob > 0.5: return 1.0else: return 0.0def colicTest():frTrain = open('horseColicTraining.txt'); frTest = open('horseColicTest.txt')trainingSet = []; trainingLabels = []for line in frTrain.readlines():currLine = line.strip().split('\t')lineArr =[]for i in range(21):lineArr.append(float(currLine[i]))trainingSet.append(lineArr)trainingLabels.append(float(currLine[21]))trainWeights = stocGradAscent1(array(trainingSet), trainingLabels, 1000)errorCount = 0; numTestVec = 0.0for line in frTest.readlines():numTestVec += 1.0currLine = line.strip().split('\t')lineArr =[]for i in range(21):lineArr.append(float(currLine[i]))if int(classifyVector(array(lineArr), trainWeights))!= int(currLine[21]):errorCount += 1errorRate = (float(errorCount)/numTestVec)print "the error rate of this test is: %f" % errorRatereturn errorRatedef multiTest():numTests = 10; errorSum=0.0for k in range(numTests):errorSum += colicTest()print "after %d iterations the average error rate is: %f" % (numTests, errorSum/float(numTests))multiTest()
结果:错误率35.8%…

