莱阳网站建设/排行榜123网
单变量过滤方法主要是基于特征变量和目标变量之间的相关性、互信息等计算出来的,总结如下:
1、最简单的方差选择法
from sklearn.feature_selection import VarianceThreshold
result=VarianceThreshold(threshold=0.5).fit_transform(data.data)
#然后根据各个特征的方差的结果来删除方差太小的特征
#如果嫌人工看麻烦可以使用selectkbest或者selectprecentile来自动选择这种方法虽然简单,但是最大的问题是将特征的重要性完全归结于统计学上的方差,然而问题在于在实际的业务场景中,可能方差很小的特征携带了非常重要的信息。举个例子,比如正负样本非常不均衡的二分类问题中,正样本有10个,负样本有10000个,我们的建模目标是尽量用模型将这10个正样本分辨出来,假设我们存在某个特征恰好正样本在该特征上取值为1,负样本在该特征上取值为0,则这个特征的方差会很小,但是确具有重要的意义,另外,方差的计算还会受到异常值的影响所以使用前可能还需要事先对异常值进行相应的处理。所以,个人一般倾向于使用这种方法来筛选方差为0或者极其接近于0的特征。
2、覆盖率
from《美团机器学习实战》,主要是针对类别型特征来计算的一个衡量指标,假设样本个数一共有10000个,某个类别特征f1一共有“A”,“B”,“C”三种,并且分别有8000个“A”,1950个“B”以及50个“C”,则“A”,“B”,“C”的覆盖率分别为:8000/10000,1950/10000,50/10000。类似的,覆盖率小的特征更容易被剔除。
#假设f1为类别型特征则:
from collections import Counter
Counter(f1) #即可计算出不同的类别特征的数量,然后分别除以总样本数量即可这个其实简单说就是根据类别特征中各个子类别的数量来进行处理,比如100000个样本某些类别出现的次数很小例如就5次或者10次,这这些出现次数很少的类别可以合并为“other”类,这样进行onehot展开的时候一方面能降低高基数类别特征的onehot展开之后维度太高的问题,一方面能够降低过拟合的风险。是高基类类别特征的一种比较常规常见而且简单好理解的处理方式,问题在于到底出现多少次算是“少数类”要合并到“other”中去?这个目前没有什么明确的标准,个人经验是根据分布图的情况来人工尝试划分几次然后比较最终的模型评价的结果。
3、互信息
简单回归一下信息论的知识:
信息奠基人香农(Shannon)认为“信息是用来消除随机不确定性的东西”。也就是说衡量信息量大小就看这个信息消除不确定性的程度。(习惯上对数的底数我们取2或者是e,下面是2的情况)
1、信息量
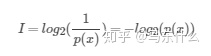
很好理解,“太阳每天从东方升起”这个事件的概率为1,则信息量I=0,因为这是一件确定性的事件,所以不确定性为0,则信息量为0。
2、信息熵
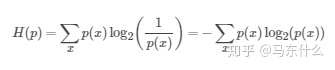
看公式,以扔硬币为例子,假设硬币均匀正反改为均为0.5,则扔均匀硬币根据公式可得信息熵就是-(1/2log2(1/2)+1/2*log2(1/2),其实就是加权平均信息量。这个和id3决策树里面用的信息熵是一个东西。信息熵用来衡量事物不确定性。信息熵越大,事物越具不确定性,事物越复杂,具有的总的信息量越大。
3、联合熵与条件熵
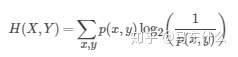
上面是联合熵的公式,其实如果把(x,y)当做一个整体z,其形式和信息熵的公式是差不多的。
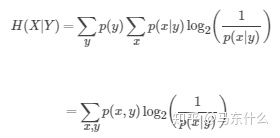
上面是条件熵,含义很直观,就是已知Y的情况下,X的信息熵的大小,举一个极端的例子,如果X和Y相互独立,则以Y为先验的X的联合熵在数值上等于X的信息熵。联合熵和信息熵的关系为:
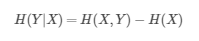
4、互信息(信息增益)
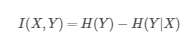
从公式来看,互信息就是衡量引入X之后,Y的信息熵的变化量,如果信息熵变化很大,说明X的引入让Y的不确定性提高了,那么就说明这个特征X相对于target Y是很重要的。
这个其实就是ID3用到的信息增益,对,两个是一个东西。
下面详细展开:
https://www.douban.com/note/621588501/ 发现了一篇关于互信息的讨论很深刻的文章,作者真是非常细心和专业!下面的代码部分就是直接根据这个网址里的内容写的
正式地,两个离散随机变量 X 和 Y 的互信息可以定义为:
其中 p(x,y) 是 X 和 Y 的联合概率分布函数,而p(x)和p(y)分别是 X 和 Y 的边缘概率分布函数。
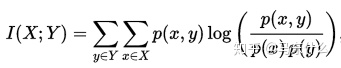
在连续随机变量的情形下,求和被替换成了二重定积分:
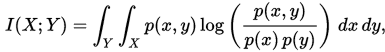
其中 p(x,y) 当前是 X 和 Y 的联合概率密度函数,而p(x)和p(y)分别是 X 和 Y 的边缘概率密度函数。但是尴尬的是,“ X 和 Y 的联合概率密度函数”以及“ X 和 Y 的边缘概率密度函数”我们事先是不知道的,它不像两个变量都是离散变量的情况那样可以直接通过计数的方式进行计算,所以更常见的是对离散的特征和目标值来计算互信息的值(当然可以对连续值进行分箱离散化之后计算互信息,决策树之所以能对连续特征进行互信息-信息增益的计算,实际上是一开始做了二分类,也就是把原来的连续特征离散化成两块称为离散型特征,所以本质上也是计算两个离散的列之间(离散化特征与分类标签)的互信息)。
from sklearn.metrics import mutual_info_score
signals=[1,1,0,0,0]
label_1=['a','a','s','s','s']
mutual_info_score(signals,label_1)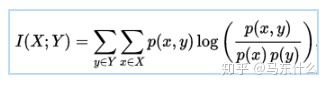
为了验证这个函数没写错还是自己手动算一下有没有问题:
0.4*math.log(2.5,np.e)+0.6*math.log(5/3.0,np.e)=0.6730116670092565 和调包的结果一样,nice。看来是没问题了。
然而接下来出现了这么一个问题
label_2=['a','b','c','d','e']
mutual_info_score(signals,label_1)计算得到的结果居然和上一段代码的结果完全一样?
从文氏图的角度出发:
首先互信息的另一个公式:
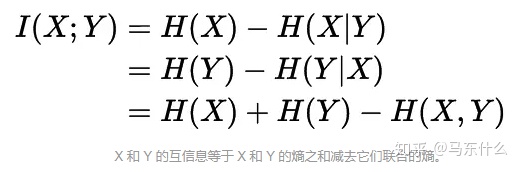
这个公式的形式直接暗示着集合的文氏图,如下:
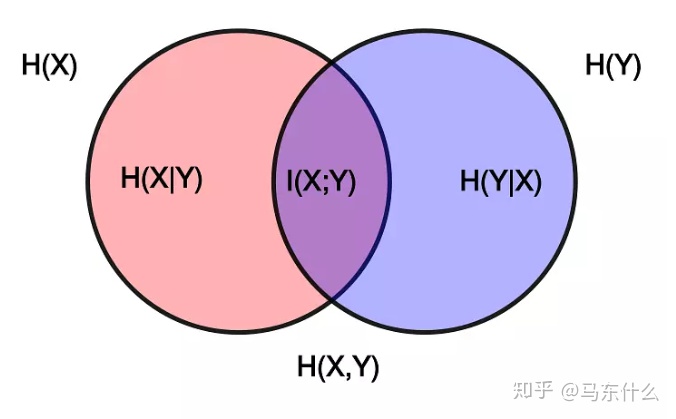
则根据互信息我们可以写出如下代码:
from collections import Counter
signals=[1,1,0,0,0]
labels_1=['a','a','s','s','s']
sig_label_1=['1a','1a','0s','0s','0s']
def entropy(labels):prob_dict=Counter(labels)s=sum(prob_dict.values())probs=np.array([i/s for i in prob_dict.values])return -prob.dot(np.log(probs))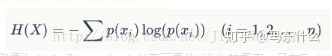
结果和上面的是完全相同的。
问题在于,更复杂的 labels_2 的熵确实变大了。但联合熵也同步变大,求差之后对消,于是交集部分的互信息毫无差异。用图来看会更明确:
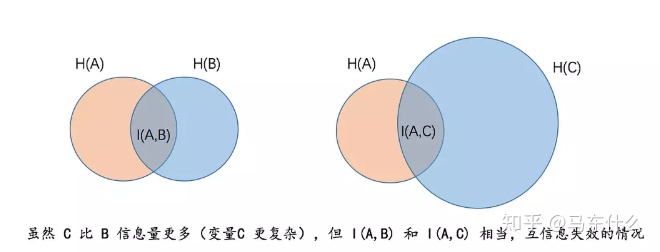
那么为了解决这个问题,就引入了标准互信息化NMI,正好sklearn中也已经做了实现。
NMI的公式如下:
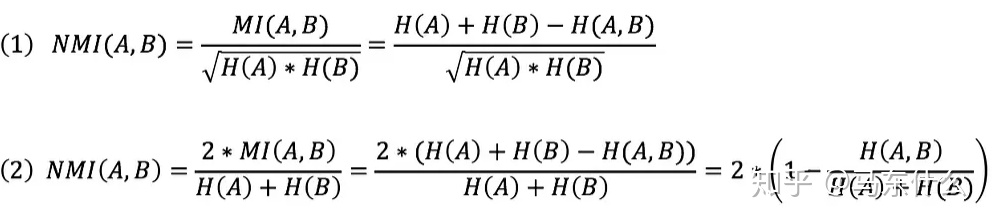
式(1)使用几何平均描述 H(A)和H(B)的规模,式(2)使用算术平均。新版的sklearn中默认使用的是算是平均的版本也就是(1)式。(标准化的计算方式有'min','geometric','arithmetic'和'max'四种,不设置则用默认的几何平均,不过官方文档的警告提示“The behavior of NMI will change in version 0.22. To match the behavior of 'v_measure_score', NMI will use average_method='arithmetic' by default.”)
然后我们重新计算一下:
from sklearn.metrics import normalized_mutual_info_score
signals=[1,1,0,0,0]
label_1=['a','a','s','s','s']
print(normalized_mutual_info_score(signals,label_1))
label_2=['a','b','c','d','e']
print(normalized_mutual_info_score(signals,label_2))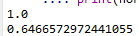
很好,总算是working了。通过标准化互信息的方式就能比较好的对不同的特征进行比较,避免了未标准化信息在上文中提到的bug。当然,即使是标准化互信息计算出来的结果也并不是放之四海而皆准的,和所有的评价指标一样,它只是从某个角度反应了特征的重要性得分而已。
4、卡方检验
(from 百度百科)卡方检验就是统计样本的实际观测值与理论推断值之间的偏离程度,实际观测值与理论推断值之间的偏离程度就决定卡方值的大小,如果卡方值越大,二者偏差程度越大;反之,二者偏差越小;若两个值完全相等时,卡方值就为0,表明理论值完全符合。
注意:卡方检验针对分类变量。
sklearn中的chi2计算的结果是卡方统计量。
源代码可查:
X = check_array(X, accept_sparse='csr')if np.any((X.data if issparse(X) else X) < 0):raise ValueError("Input X must be non-negative.")Y = LabelBinarizer().fit_transform(y)if Y.shape[1] == 1:Y = np.append(1 - Y, Y, axis=1)observed = safe_sparse_dot(Y.T, X) # n_classes * n_featuresfeature_count = X.sum(axis=0).reshape(1, -1)class_prob = Y.mean(axis=0).reshape(1, -1)expected = np.dot(class_prob.T, feature_count)https://blog.csdn.net/ludan_xia/article/details/81737669 找到了一篇解释的比较清楚的关于卡方统计量计算的文章:
为了验证sklearn的chi2的计算公式没问题(懒得去折腾源代码了直接算一算比较一下就行),使用一些简单数据手动计算结果然后再用sklearn的chi2计算比较:
import numpy as npdrink_milk=np.ones(139) #139个喝牛奶的
target1=drink_milk.copy()
target1[43:139]=0 #96个人没感冒,43个人感冒
not_drink_milk=np.zeros(112)
target2=not_drink_milk.copy()
target2[0:28]=1 x=np.concatenate([drink_milk,not_drink_milk])
y=np.concatenate([target1,target2])from sklearn.feature_selection import chi2
chi2(x,y)结果发现。。。怎么计算的结果不一样???
def _chisquare(f_obs, f_exp):"""Fast replacement for scipy.stats.chisquare.Version from https://github.com/scipy/scipy/pull/2525 with additionaloptimizations."""f_obs = np.asarray(f_obs, dtype=np.float64)k = len(f_obs)# Reuse f_obs for chi-squared statisticschisq = f_obschisq -= f_expchisq **= 2with np.errstate(invalid="ignore"):chisq /= f_expchisq = chisq.sum(axis=0)return chisq, special.chdtrc(k - 1, chisq)def chi2(X, y):"""Compute chi-squared stats between each non-negative feature and class.This score can be used to select the n_features features with thehighest values for the test chi-squared statistic from X, which mustcontain only non-negative features such as booleans or frequencies(e.g., term counts in document classification), relative to the classes.Recall that the chi-square test measures dependence between stochasticvariables, so using this function "weeds out" the features that are themost likely to be independent of class and therefore irrelevant forclassification.Read more in the :ref:`User Guide <univariate_feature_selection>`.Parameters----------X : {array-like, sparse matrix}, shape = (n_samples, n_features_in)Sample vectors.y : array-like, shape = (n_samples,)Target vector (class labels).Returns-------chi2 : array, shape = (n_features,)chi2 statistics of each feature.pval : array, shape = (n_features,)p-values of each feature.Notes-----Complexity of this algorithm is O(n_classes * n_features).See also--------f_classif: ANOVA F-value between label/feature for classification tasks.f_regression: F-value between label/feature for regression tasks."""# XXX: we might want to do some of the following in logspace instead for# numerical stability.X = check_array(X, accept_sparse='csr')if np.any((X.data if issparse(X) else X) < 0):raise ValueError("Input X must be non-negative.")Y = LabelBinarizer().fit_transform(y)if Y.shape[1] == 1:Y = np.append(1 - Y, Y, axis=1)observed = safe_sparse_dot(Y.T, X) # n_classes * n_featuresfeature_count = X.sum(axis=0).reshape(1, -1)class_prob = Y.mean(axis=0).reshape(1, -1)expected = np.dot(class_prob.T, feature_count)return _chisquare(observed, expected)
测了半天终于找到问题了。。。真是服了,chi2仅仅计算y=1的时候的卡方值,对照原文:


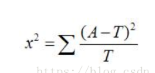
按照公式来看的话应该是:(43-39.3231)**2/39.3231+(96-99.6769)**2/99.6769+(28-31.6848)**2/31.6848+(84-80.3125)**2/80.3125=1.077和手算的结果是一样的,但是根据chi2的源代码显示,它只计算了(43-39.3231)**2/39.3231+(96-99.6769)**2/99.6769=0.4806。。。。。。也就是仅仅计算了喝牛奶组的情况(如上图),不喝牛奶组的计算结果没有计入在内。。。。wadu heck
好吧,只能说算是计算了特征相对于目标值“1”的卡方值。
折腾死了、、、、、、
5、Fisher得分
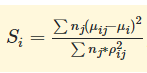
其中Uij和Pij分别为特征i在类别j中的均值和方差,Ui为特征i的均值,nj为类别j中的样本数量。所以显然,fisher scoring针对的是连续型的feature与离散型的target。feature在不同的类别target之间的差异越大,在同一个类别中的差异越小,则特征越重要。
非常nice,居然找到了skfeature这种东西。。。
https://github.com/jundongl/scikit-feature git下载下来然后python setup.py install,pip 没法下载,应该是都没放到pypy里
#源代码:
def fisher_score(X, y):"""This function implements the fisher score feature selection, steps are as follows:1. Construct the affinity matrix W in fisher score way2. For the r-th feature, we define fr = X(:,r), D = diag(W*ones), ones = [1,...,1]', L = D - W3. Let fr_hat = fr - (fr'*D*ones)*ones/(ones'*D*ones)4. Fisher score for the r-th feature is score = (fr_hat'*D*fr_hat)/(fr_hat'*L*fr_hat)-1Input-----X: {numpy array}, shape (n_samples, n_features)input datay: {numpy array}, shape (n_samples,)input class labelsOutput------score: {numpy array}, shape (n_features,)fisher score for each featureReference---------He, Xiaofei et al. "Laplacian Score for Feature Selection." NIPS 2005.Duda, Richard et al. "Pattern classification." John Wiley & Sons, 2012."""# Construct weight matrix W in a fisherScore waykwargs = {"neighbor_mode": "supervised", "fisher_score": True, 'y': y}W = construct_W(X, **kwargs)# build the diagonal D matrix from affinity matrix WD = np.array(W.sum(axis=1))L = Wtmp = np.dot(np.transpose(D), X)D = diags(np.transpose(D), [0])Xt = np.transpose(X)t1 = np.transpose(np.dot(Xt, D.todense()))t2 = np.transpose(np.dot(Xt, L.todense()))# compute the numerator of LrD_prime = np.sum(np.multiply(t1, X), 0) - np.multiply(tmp, tmp)/D.sum()# compute the denominator of LrL_prime = np.sum(np.multiply(t2, X), 0) - np.multiply(tmp, tmp)/D.sum()# avoid the denominator of Lr to be 0D_prime[D_prime < 1e-12] = 10000lap_score = 1 - np.array(np.multiply(L_prime, 1/D_prime))[0, :]# compute fisher score from laplacian score, where fisher_score = 1/lap_score - 1score = 1.0/lap_score - 1return np.transpose(score)惊喜的发现fisher_score 实现是纯函数式的而且用的numpy,这样用后期如果需要提高运行速度可以比较方便的用numba或者cython来做加速
from skfeature.function.similarity_based import fisher_score
from sklearn.datasets import load_iris
X=load_iris().data
y=load_iris().target
print(fisher_score.fisher_score(X,y))6、woe编码和IV值
懒得手打,下文来自于(https://www.cnblogs.com/WoLykos/p/9584751.html)
woe编码和IV值的计算主要针对的是离散型特征和二分类问题所提出的一种特征重要性的衡量方法,在信贷、风控模型中很常见,iv是在woe的基础上计算的,所以我们先简单了解一下woe的概念。
woe全称是“Weight of Evidence”,即证据权重,是对原始自变量的一种编码形式。
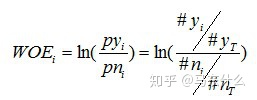
进行WOE编码前,需要先把这个变量进行分组处理(离散化)
其中,pyi是这个组中响应客户(即模型中预测变量取值为“是”或1的个体,也叫坏样本)占所有样本中所有响应客户的比例,pni是这个组中未响应客户(也叫好样本)占样本中所有未响应客户的比例;
#yi是这个组中响应客户的数量,#ni是这个组中未响应客户的数量,#yT是样本中所有响应客户的数量,#nT是样本中所有未响应客户的数量。
从这个公式中我们可以体会到,WOE表示的实际上是“当前分组中响应客户占所有响应客户的比例”和“当前分组中没有响应的客户占所有没有响应的客户的比例”的差异。
为了更简单明了一点,我们来做个简单变换,得:
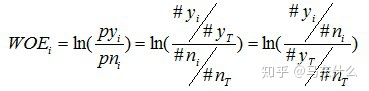
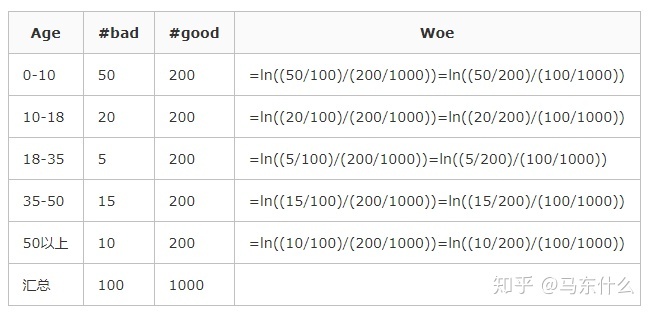
不难看出,woe表示的是当前这个组中响应客户和未响应客户的比值,和所有样本中这个比值的差异。这个差异是用这两个比值的比值,再取对数来表示的。例子如下:
WOE越大,这种差异越大,这个分组里的样本响应的可能性就越大,WOE越小,差异越小,这个分组里的样本响应的可能性就越小。woe反映的是在自变量每个分组下违约用户对正常用户占比和总体中违约用户对正常用户占比之间的差异;从而可以直观的认为woe蕴含了自变量取值对于目标变量(违约概率)的影响。再加上woe计算形式与logistic回归中目标变量的logistic转换(logist_p=ln(p/1-p))如此相似,因而可以将自变量woe值替代原先的自变量值。(注意,所谓woe编码就是将某个类别特征的各个类别分别用它的woe值进行替换,这样就解决了类别变量展开之后的稀疏问题。)
woe的手动编写就不用我写了吧,网上一大堆自己找一个就行了,计算思路也很简单,下面推荐的是一个很nice的scikit_learncontrib——categorical_encoders,最新版本的还支持catboost对分类变量的编码方式,cool,代码本身逻辑也比较easy,后期如果需要自主修改也比较方便。(根据源代码的提示,对于woe编码中可能存在的除0问题,woeencoder默认是使用1来代替除数,这个参数的大小可以自己设置,同时对于训练集中存在的类别如果测试集中出现了新的则使用handle_unknown来处理,具体参看官网的api介绍)
from category_encoders.woe import WOEEncoder
woe=WOEEncoder(cols=cols,handle_unknown='return_nan')
woe_result=woe.fit_transform(data[cols],target)具体用法参考官方文档就行了。
对于一个分组后的变量,第i 组的WOE前面已经介绍过,同样,对于分组i,也会有一个对应的IV值,计算公式如下:
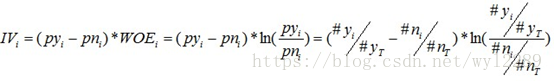
有了一个变量各分组的IV值,我们就可以计算整个变量的IV值,方法很简单,就是把各分组的IV相加:
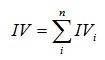
这样我们就最终得到了每一个特征对应的IV值。至于为什么我们不使用woe的绝对值相加的方式而不通过IV这种类似加权的方式来计算结果的方式的原因在于,如果某一类出现的比例占比很小可能会导致其woe的值很大从而影响判断,具体的原因可以参看下面的链接,不赘述。
https://blog.csdn.net/iModel/article/details/79420437blog.csdn.net从woe的计算公式可以看出,WOE其实描述了变量当前这个分组,对判断个体是否会响应(或者说属于哪个类)所起到影响方向和大小,当WOE为正时,变量当前取值对判断个体是否会响应起到的正向的影响,当WOE为负时,起到了负向影响。而WOE值的大小,则是这个影响的大小的体现。为了规避不同类别的比例大小对woe计算的影响从而引入了IV值的计算,所以IV值越大则表示这个特征对于正负样本的区分的贡献度越大。
7、相关系数
严格来说,相关系数有三种,pearson、spearman、kendall,我们平常最常说最常用的是pearson皮尔森相关系数,公式如下:
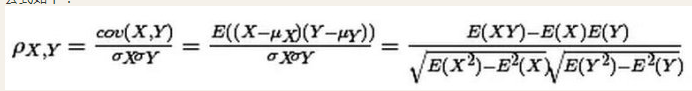
需要注意的是如果两个变量中有一个为完全相同的特征,即特征的取值都是同一个值,那么相关系数是计算不出来的(不过这种特征一般都会删掉吧。。。)
通过公式可以看出,计算相关系数的时候是不需要进行标准化的:
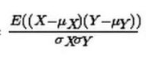
相关系数的计算过程中本身就包含了一个标准化的过程从而消除了不同量纲的影响。
简单的相关系数的分类
- 0.8-1.0 极强相关
- 0.6-0.8 强相关
- 0.4-0.6 中等程度相关
- 0.2-0.4 弱相关
- 0.0-0.2 极弱相关或无相关
但是pearson相关系数有一个比较麻烦的问题:
1、 它对于线性关系敏感对于非线性关系不敏感,非线性关系即使是一一对应的关系使用pearson相关系数计算出来的结果也会比较差。比如x1=【-5,-3,-1,1,3,5】而x2=x1**2=【25,9,1,1,9,25】,二者之间是一一对应的关系,但是pearson相关系数为-0.5多
2、对异常值很敏感。这个看公式就知道了,自己手动算一下也可以。
一般来说,使用线性回归和逻辑回归的时候会使用相关系数作为特征重要性的评价指标之一,而如果是树模型则更倾向于使用下面的这个相关系数。
spearman 秩相关系数
懒得写了直接上网址:
https://blog.csdn.net/qq_30138291/article/details/79801777blog.csdn.net斯皮尔曼相关性系数,通常也叫斯皮尔曼秩相关系数。“秩”,可以理解成就是一种顺序或者排序,那么它就是根据原始数据的排序位置进行求解,这种表征形式就没有了求皮尔森相关性系数时那些限制。下面来看一下它的计算公式:
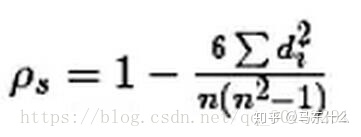
计算过程就是:首先对两个变量(X, Y)的数据进行排序,然后记下排序以后的位置(X’, Y’),(X’, Y’)的值就称为秩次,秩次的差值就是上面公式中的di,n就是变量中数据的个数,最后带入公式就可求解结果。
很显然,spearm对于异常值一点也不敏感。并且它表示的是两列数据之间的排序的相关性。如果使用树模型,spearman进行特征选择会更合理一些。
8、sklearn中还存在着这几种过滤式评价指标
简单看了一下理论推导还是比较复杂的,鉴于使用的并不多,所以,心情好的时候再吧。。。
至于mRMR,emmm,其实和相关性的选择方法存在一样的缺陷,就是无法考虑到原始特征较弱而组合特征较强的情况(比如目标变量由特征变量进行异或得到的情况),所以我就不赘述了,至于《美团机器学习实践》中提到的CFS实际上不能算是严格意义上的过滤式特征选择方法,他的原理应该划入包裹式特征消除方法的范畴中,所以后续写到这里的时候再详细展开吧。
